Cet article est la traduction intégrale par Google des informations fournies par le comité Nobel, à l’occasion de la remise du prix Nobel de physiologie ou de médecine à Katalin Karikó et Drew Weissman. Il faut consulter l’article original pour voir les références.
Lorsque le SARS-CoV-2 a émergé fin 2019 et s’est rapidement propagé à travers le monde, rares étaient ceux qui pensaient que des vaccins pourraient être développés à temps pour freiner la progression de la maladie à l’échelle mondiale. Pourtant, plusieurs vaccins ont été approuvés en un temps record, dont deux des vaccins les plus rapidement approuvés et les plus efficaces, produits grâce à la nouvelle technologie de l’ARNm. Le concept d’utilisation de l’ARNm pour la vaccination et l’administration in vivo de protéines thérapeutiques a été proposé pour la première fois il y a plus de 30 ans, mais de nombreux obstacles ont dû être surmontés avant de devenir une réalité clinique. Les premières expériences ont démontré que l’ARNm transcrit in vitro stimulait des réponses inflammatoires indésirables et une production protéique inefficace dans les cellules et les tissus. Un tournant décisif a été la découverte de Karikó et Weissman démontrant que l’ARNm produit avec des bases nucléosidiques modifiées échappe à la reconnaissance immunitaire innée et améliore l’expression protéique. Ces découvertes, conjuguées à la mise au point de systèmes efficaces d’administration d’ARNm in vivo, à la stabilisation de l’antigène Spike du SARS-CoV-2 et à des investissements sans précédent de l’industrie et des gouvernements, ont permis l’approbation de deux vaccins contre la COVID-19 à base d’ARNm très performants fin 2020. La découverte de Karikó et Weissman a été cruciale pour rendre la plateforme vaccinale à ARNm utilisable en clinique au moment où elle était le plus nécessaire, constituant ainsi une contribution extraordinaire à la médecine et ouvrant la voie à de futures applications de l’ARNm.
Dans notre société mondialisée et interconnectée, le risque de nouvelles pandémies est plus élevé que jamais. Les pandémies sont généralement causées par des virus zoonotiques qui franchissent la barrière des espèces et se transmettent à l’homme par voie aérienne (gouttelettes ou aérosols), provoquant des infections respiratoires. Développer et déployer des vaccins suffisamment rapidement pour atténuer une pandémie en cours représente un défi colossal, inédit avant la pandémie de COVID-19. Le partage rapide de la séquence du génome du SARS-CoV-2, conjugué aux progrès considérables réalisés ces dernières décennies en biologie moléculaire, en recherche vaccinale et en administration de médicaments, a stimulé une activité sans précédent chez les chercheurs en vaccins en 2020. Des scientifiques du monde universitaire et de l’industrie ont lancé des projets en un temps record, bénéficiant du soutien financier et logistique des gouvernements, de l’industrie et d’organisations à but non lucratif. La nouvelle plateforme vaccinale à ARNm représentait l’une des options les plus prometteuses, mais son efficacité contre ce nouveau virus restait inconnue. Aucun vaccin à ARNm n’avait été approuvé pour l’usage humain auparavant.
Plateformes de vaccins viraux avant la COVID-19
La plupart des vaccins antiviraux homologués actuellement disponibles sont produits selon des techniques traditionnelles à partir de virus entiers atténués ou inactivés (Figure 1). Les vaccins vivants atténués, tels que le vaccin combiné rubéole-oreillons-rougeole et le vaccin contre la fièvre jaune, induisent une immunité humorale et cellulaire robuste et durable. Pour la mise au point du vaccin contre la fièvre jaune, Max Theiler a reçu le prix Nobel de physiologie ou médecine en 1951. Les vaccins à base de virus inactivés, tels que le vaccin contre l’encéphalite à tiques et le vaccin contre l’hépatite A, induisent des réponses immunitaires efficaces mais plus transitoires, nécessitant des rappels réguliers. La révolution de la biologie moléculaire et le développement des technologies de production de protéines recombinantes ont ouvert la voie à des approches vaccinales plus ciblées. Le premier vaccin produit selon cette approche fut le vaccin contre l’hépatite B (VHB), approuvé en 1986, suivi en 2006 par le premier vaccin contre le papillomavirus humain (VPH). Les vaccins contre le VHB et le VPH contiennent des composants protéiques uniques du virus respectif et sont appelés vaccins sous-unitaires. Ces vaccins protègent contre les cancers induits par les virus et constituent une avancée majeure [1]. Les progrès de la biologie moléculaire ont également permis la conception de virus vecteurs codant pour des antigènes hétérologues d’intérêt. Ces vecteurs viraux pénètrent efficacement dans les cellules où les antigènes codés sont produits par la machinerie de synthèse protéique endogène. Le premier exemple de vaccin à vecteur viral homologué fut le vaccin contre Ebola à base de virus de la stomatite vésiculaire, approuvé en 2019, rapidement suivi par un vaccin contre Ebola à base d’adénovirus [2].
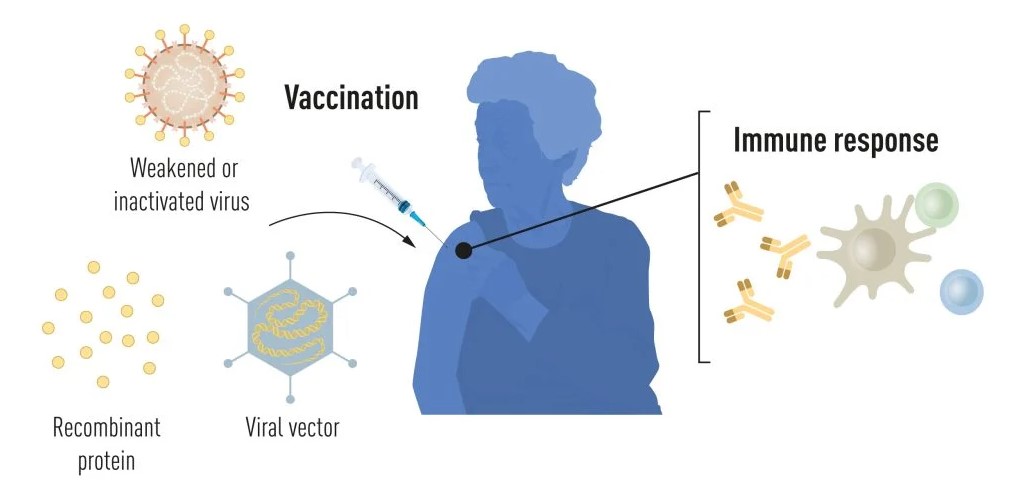
Les vaccins actuellement utilisés sont fabriqués à partir de virus entiers atténués ou inactivés, de composants protéiques viraux recombinants (vaccins sous-unitaires) ou de vecteurs viraux délivrant les antigènes d’intérêt (vaccins à vecteur). La vaccination stimule des réponses immunitaires spécifiques à l’antigène, offrant une protection en cas d’exposition ultérieure au pathogène vivant. © Comité Nobel de physiologie ou médecine. Ill. Mattias Karlén
Les vaccins traditionnels à base de virus entier et les vaccins à vecteur viral nécessitent tous deux des installations de production basées sur la culture cellulaire. C’est pourquoi les chercheurs en vaccinologie s’intéressent depuis longtemps au développement de vaccins sous-unitaires qui s’affranchissent du recours aux cultures cellulaires à grande échelle en administrant directement l’acide nucléique (ADN ou ARNm) aux personnes vaccinées, exploitant ainsi la capacité de l’organisme à produire des protéines. On pensait généralement que la disponibilité de telles plateformes permettrait non seulement d’accroître la capacité mondiale de production de vaccins, mais aussi de faciliter une production plus rapide et moins coûteuse en cas de pandémie.
Premiers travaux sur les vaccins à base d’acide nucléique et à vecteur viral
Les premières démonstrations de l’efficacité des immunisations à base d’acide nucléique remontent au début des années 1990, lorsque des vaccins à ADN [3] et à ARNm [4] ont été testés pour la première fois chez la souris. Ces approches présentaient plusieurs avantages potentiels. Non seulement les vaccins à base d’acide nucléique sont faciles à produire, mais ils sont également flexibles, car leur séquence peut être facilement modifiée pour coder différents antigènes. Conjuguée à la facilité de production, cette caractéristique rend les tests itératifs de nouveaux vaccins candidats et la génération de vaccins améliorés rapides et efficaces. Un avantage biologique réside dans le fait que, outre les réponses des lymphocytes T CD4+ [auxiliaires] restreintes par les anticorps et le complexe majeur d’histocompatibilité (CMH) de classe II, également induites par d’autres types de vaccins, les vaccins à vecteur viral et à base d’acides nucléiques ont le potentiel de stimuler les réponses des lymphocytes T CD8+ cytotoxiques, car ils permettent la présentation de peptides antigéniques endogènes sur les molécules du CMH de classe I. L’induction des lymphocytes T CD8+ est particulièrement intéressante dans le contexte des vaccins anticancéreux, dont l’objectif est de détruire les cellules tumorales ciblées, ainsi que pour les vaccins antiviraux visant à éliminer les cellules infectées. Cependant, malgré les avantages potentiels des vaccins à base d’acides nucléiques, leur tolérance et leur capacité à stimuler une réponse immunitaire suffisamment robuste chez l’homme pour constituer une voie viable pour le développement clinique des vaccins restaient incertaines.
Initialement, les vaccins à ADN étaient considérés comme plus prometteurs que les vaccins à ARNm, l’ADN étant plus stable. Cependant, les progrès ont été lents et les premiers résultats encourageants obtenus avec les vaccins à ADN chez les petits animaux ne se sont pas transposés à l’homme [5]. Une explication probable réside dans le fait que l’ADN injecté doit franchir deux barrières, la membrane plasmique et la membrane nucléaire, pour atteindre le compartiment cellulaire où a lieu la transcription (conversion de l’ADN en ARNm). À l’inverse, les vaccins à ARNm n’ont besoin d’accéder qu’au cytoplasme cellulaire où se déroule la traduction (conversion de l’ARNm en protéine), ce qui facilite leur administration. Un autre avantage des vaccins à ARNm est que l’acide nucléique administré ne peut s’intégrer au génome de l’hôte, ce qui constitue un atout majeur en matière de sécurité. Malgré ces avantages, le scepticisme quant à l’utilité de cette approche est resté important, l’ARNm étant jugé trop instable pour les applications médicales.
Dans ce contexte, le domaine de la vaccination s’est tourné vers l’utilisation de vecteurs viraux modifiés, ceux-ci possédant leurs propres mécanismes intrinsèques pour pénétrer dans les cellules et y délivrer leur charge génétique. Depuis les années 1990, de nombreux vaccins à vecteur viral ciblant divers pathogènes ont été testés en préclinique, révélant des résultats prometteurs mais aussi des échecs [6]. Un inconvénient de ces vaccins réside dans le risque d’induction d’anticorps contre les protéines structurales utilisées pour l’encapsulation du vecteur, en plus des réponses immunitaires recherchées contre l’antigène ciblé. Ceci peut compromettre l’efficacité des rappels vaccinaux en cas de réutilisation du même vecteur. Néanmoins, des vaccins à vecteur viral efficaces, utilisant différents types d’adénovirus modifiés, ont été développés pendant la pandémie de COVID-19 et administrés à grande échelle, démontrant leur utilité, notamment en phase initiale de pandémie [7, 8].
Au cours des années 1990, une petite équipe de chercheurs a continué d’explorer l’utilisation de l’ARNm comme plateforme vaccinale potentielle. Les premières études avaient démontré que l’ARNm purifié à partir de cellules était traduit en protéine après réintroduction dans des ovocytes [9]. L’étape suivante consistait à acheminer le vaccin vers les tissus d’un organisme vivant. La première étude démontrant que l’injection d’ARNm nu dans le muscle squelettique entraînait la production de protéines in vivo a été publiée par Philip Felgner et ses collègues en 1990 [10]. Peu après, Martinon et al. ont démontré l’induction de réponses des lymphocytes T cytotoxiques spécifiques de l’antigène chez des souris ayant reçu une injection d’ARNm liposomal codant pour la nucléoprotéine du virus de la grippe [4].
Parallèlement, plusieurs chercheurs ont développé des vaccins à réplicons d’alphavirus, qui présentent l’avantage supplémentaire de produire un plus grand nombre de copies de transcrits codant pour l’antigène dans chaque cellule, induisant ainsi de robustes réponses immunitaires spécifiques de l’antigène après administration in vivo d’ARNm nu [11, 12]. Ces premières études ont stimulé la recherche et ont permis d’obtenir des résultats prometteurs chez l’animal, mais il faudra attendre plus de vingt ans avant que le premier vaccin à ARNm contre une infection ne soit testé lors d’essais cliniques chez l’humain.
La découverte de l’ARN messager et des systèmes de transcription in vitro
Pour explorer le potentiel des applications basées sur l’ARNm, un système efficace de production et de manipulation de l’ARNm était nécessaire. Ce domaine s’est appuyé sur une série de découvertes fondamentales initiées dans les années 1950. Après la découverte majeure de l’ADN comme support de l’hérédité, la recherche a débuté pour identifier la molécule intermédiaire transcrite à partir de l’ADN nucléaire et transportée jusqu’aux ribosomes dans le cytoplasme afin de spécifier la synthèse protéique. Des expériences sur des cellules infectées par le bactériophage T2 ont permis d’identifier une fraction d’ARN métaboliquement active, représentant environ 1 % de l’ARN cellulaire total [13] et présentant des proportions de bases adéquates [14]. Cette forme instable d’ARN, ou ARN messager (ARNm), a été proposée comme le vecteur d’information intermédiaire manquant [15], et cette hypothèse a rapidement été validée expérimentalement par des expériences de marquage par impulsion chez les bactéries [16, 17]. À peu près à la même époque, la découverte de l’ARN polymérase [18-20] a permis de mieux comprendre comment les cellules produisent de l’ARN à partir de l’ADN. Au cours des décennies suivantes, plusieurs ARN polymérases ont été identifiés chez les bactéries et les cellules eucaryotes, notamment des ARN polymérases à une seule sous-unité issus des bactériophages T7 [21] et SP6 [22].
S’appuyant sur la découverte des ARN polymérases de bactériophages, plus polyvalente, Paul Krieg et Douglas Melton ont démontré qu’il était possible de produire de l’ARNm synthétique en grande quantité in vitro grâce à l’ARN polymérase SP6 et à des clones d’ADNc contenant le promoteur SP6 [23, 24]. De plus, l’ARNm SP6 produit in vitro a été efficacement traduit en protéine après injection dans des ovocytes de grenouille [23]. À peu près à la même époque, l’ARN polymérase T7 a été cloné par le laboratoire de William Studier [25] et développé en un système de transcription in vitro efficace et inductible, breveté en 1984 [26]. L’ARN polymérase T7 présentait plusieurs avantages, notamment une liaison très spécifique au promoteur T7 (une séquence conservée de nucléotides -17 à +6 par rapport au site d’initiation de la transcription) et une capacité à transcrire l’ARN rapidement. Des efforts similaires visant à exploiter le potentiel de transcription in vitro de l’ARN polymérase T7 ont été entrepris [27]. Le système de transcription in vitro T7 a ensuite été optimisé pour devenir un système acellulaire très efficace permettant la production à grande échelle de tout ARNm d’intérêt, avec un impact majeur sur la science et la biotechnologie.
Délivrance d’ARN messager transcrit in vitro aux cellules et aux tissus
Un autre axe de recherche important s’est concentré sur la délivrance d’acides nucléiques dans les cellules. Une des premières stratégies consistait à utiliser des liposomes, de petites vésicules semblables à la membrane cellulaire, composées de phospholipides et de cholestérol. Dès 1978, des chercheurs avaient décrit des succès dans la délivrance d’ARNm de globine purifié dans des lymphocytes de souris et des cellules épithéliales humaines grâce à des liposomes [28, 29], par simple piégeage de l’ARNm à l’intérieur des vésicules liposomales. Le domaine de la délivrance d’acides nucléiques a progressé grâce aux travaux pionniers de Philip Felgner chez Syntex Research. Felgner a synthétisé le premier lipide cationique (DOTMA) et a démontré sa capacité à former des liposomes stables avec des acides nucléiques [30]. La charge positive des lipides a amélioré à la fois le piégeage des acides nucléiques chargés négativement (par interactions électrostatiques) et leur fusion aux membranes cellulaires chargées négativement, ce qui a permis une meilleure délivrance dans les cellules. Les liposomes à base de lipides cationiques (lipofectine) ont ouvert la voie à l’administration d’ADN et d’ARN modifiés dans les cellules. La lipofectine a rapidement été utilisée pour introduire de l’ARNm transcrit in vitro dans des cellules en culture afin de démontrer la production de protéines [31], encourageant ainsi de futures applications thérapeutiques. Cependant, les applications in vivo de la lipofectine ont révélé des effets secondaires indésirables, et les chercheurs ont poursuivi leurs efforts pour développer des systèmes d’administration améliorés.
Une seconde avancée majeure a été réalisée au laboratoire de Pieter Cullis à l’Université de Colombie-Britannique avec la mise au point de lipides cationiques ionisables. Ces lipides pouvaient être maintenus sous une forme chargée positivement ou neutre selon le pH du milieu. La formation de ces nanoparticules lipidiques (LNP) à pH faible présentait l’avantage, propre aux lipides cationiques, d’encapsuler efficacement l’ARNm chargé négativement au sein des vésicules. Toutefois, une fois administrées in vivo et exposées à un pH physiologique, les lipides perdaient leur charge, ce qui présentait plusieurs avantages, notamment une toxicité in vivo réduite. Les importantes découvertes de l’équipe de Cullis ont suscité un vif intérêt industriel pour le développement des lipides ionisables. Notamment, l’administration d’acides nucléiques a été optimisée grâce au connecteur en T, capable de générer des nanoparticules lipidiques denses composées de quatre éléments : i) un lipide cationique ionisable, ii) un lipide auxiliaire, iii) du cholestérol et iv) du polyéthylène glycol (PEG) [32]. Des lipides cationiques ionisables plus performants ont été identifiés lors de programmes de criblage à grande échelle menés par plusieurs entreprises de biotechnologie. Par conséquent, les nanoparticules lipidiques permettent désormais l’administration in vivo sûre et efficace d’acides nucléiques, notamment d’ARNm, dans les cellules humaines. Cette avancée est cruciale pour les applications cliniques des technologies basées sur les acides nucléiques.
Une vision visant à utiliser l’ARN messager pour l’administration de protéines thérapeutiques
Le potentiel des nouvelles techniques de biologie moléculaire pour créer des vaccins à base d’ARNm ou traiter des maladies humaines par l’administration d’ARNm afin de remplacer des gènes défectueux par des gènes fonctionnels, ou encore par la surexpression d’une protéine thérapeutique, a suscité un vif intérêt. En 1992, Jirikowski et al. ont utilisé l’injection d’ARNm pour l’expression in vivo de la vasopressine afin de traiter le diabète insipide chez un modèle murin [33]. À la même époque, une chercheuse hongroise de l’Université de Pennsylvanie, Katalin Karikó, expérimentait différentes formes d’ARN dans le but d’optimiser l’expression de protéines thérapeutiques. Karikó a obtenu son doctorat au Centre de recherche biologique de Szeged en 1982. Après un post-doctorat à l’Académie hongroise des sciences, puis des postes de chercheuse à l’Université Temple de Philadelphie et à l’Université des services de santé en uniforme de Bethesda, elle a créé son propre groupe au Département de neurochirurgie de l’Université de Pennsylvanie en 1997. Animée d’une forte volonté de faire progresser la plateforme d’ARNm, elle a étudié de manière systématique différents composants de l’ARNm transcrit in vitro afin d’identifier les conditions nécessaires à une expression protéique optimale dans les cellules et les tissus [34]. Parmi ses nombreuses découvertes, elle a démontré que l’ARNm complexé à la lipofectine et codant pour la luciférase, une protéine rapportrice, pouvait être délivré au cerveau du rat et que l’expression était améliorée par l’ajout d’une queue poly(A) plus longue à l’extrémité 3′ de l’ARNm [35]. Encouragée par ces résultats, Karikó a poursuivi ses travaux pour rendre la plateforme d’ARNm utilisable en clinique.
Transport d’ARN messager aux cellules dendritiques et rôle de la détection innée
À la fin des années 1990, Karikó s’est associé à Drew Weissman, médecin-chercheur spécialisé en immunologie fondamentale et en développement de vaccins, qui avait rejoint l’Université de Pennsylvanie en 1997. Weissman avait obtenu son doctorat en médecine et son doctorat en immunologie et microbiologie à l’Université de Boston en 1987. Après un internat au Beth Israel Deaconess Medical Center de la Harvard Medical School à Boston, il a intégré l’équipe d’Anthony Fauci aux National Institutes of Health (NIH) pour un stage postdoctoral afin d’étudier les interactions du virus de l’immunodéficience humaine de type 1 (VIH-1) avec ses récepteurs cibles sur différents types de cellules immunitaires. Ayant créé sa propre équipe à l’Université de Pennsylvanie, il s’est de plus en plus consacré à la recherche vaccinale et à l’utilisation des cellules dendritiques pour stimuler la réponse immunitaire. Ralph Steinman a reçu le prix Nobel de physiologie ou médecine en 2011 pour sa découverte des cellules dendritiques. Forts de l’expertise de Weissman en immunologie et de celle de Karikó en biochimie de l’ARN, les deux scientifiques se complétaient parfaitement et partageaient la même passion pour l’exploitation de l’ARNm en médecine.
Ensemble, Karikó et Weissman ont testé la possibilité d’introduire de l’ARNm transcrit in vitro dans les cellules dendritiques afin d’exploiter leur potentiel de présentation d’antigènes. L’un des principaux objectifs de Weissman était de développer un vaccin contre le VIH-1, un virus responsable d’infections chroniques. Ce défi était d’autant plus important que ce virus possède de nombreuses capacités d’échappement immunitaire, le distinguant des virus provoquant des infections aiguës. Weissman s’intéressait à l’utilisation des cellules dendritiques pour activer les lymphocytes T spécifiques d’un antigène et avait mis au point des systèmes permettant de cultiver ces cellules et d’évaluer leur activation et leurs capacités de présentation d’antigènes. Les cellules dendritiques possèdent une capacité remarquable à la fois à détecter les pathogènes et à activer les lymphocytes T naïfs, assurant ainsi la liaison entre les systèmes immunitaires inné et adaptatif [36]. Karikó et Weissman ont montré que des cellules dendritiques chargées d’ARNm transcrit in vitro codant pour la protéine structurale Gag du VIH-1 stimulaient les réponses primaires des lymphocytes T CD4+ et CD8+ in vitro [37]. L’équipe a également constaté que le chargement de l’ARNm entraînait l’activation et la maturation des cellules dendritiques [38], un phénomène initialement interprété comme un effet positif, les cellules dendritiques activées étant plus efficaces pour l’activation des lymphocytes T. Les conséquences négatives de l’activation de l’immunité innée par l’ARNm transcrit in vitro n’étaient pas encore pleinement comprises. De façon intéressante, et quelque peu contre-intuitive, ce phénomène s’avérera être un facteur déterminant pour le développement des vaccins à ARNm.
L’observation de l’activation des cellules dendritiques suite à l’internalisation d’ARNm transcrit in vitro a soulevé des questions cruciales quant aux voies de signalisation impliquées. Les cellules dendritiques expriment des récepteurs Toll-like (TLR) à la fois à leur surface et dans leurs endosomes. Ces récepteurs reconnaissent des structures moléculaires spécifiques appelées motifs moléculaires associés aux pathogènes (PAMP) [39]. La liaison des TLR aux PAMP induit une signalisation intracellulaire et la production de cytokines antivirales, notamment les interférons de type I, un système d’alerte efficace pour détecter les pathogènes. L’étude des mécanismes de reconnaissance des différentes formes d’acides nucléiques par les TLR a pris de l’ampleur après la découverte par Hemmi et al. que les motifs CpG non méthylés, abondants dans l’ADN microbien mais rares dans l’ADN des mammifères, activent le TLR9 [40].
En quelques années, les ligands de la plupart des TLR sensibles aux acides nucléiques ont été identifiés, notamment le TLR3, qui détecte l’ARN double brin (ARNdb), un intermédiaire de la réplication virale, ainsi que les TLR7 et TLR8, qui détectent l’ARN viral simple brin et certaines formes d’ARN synthétique [41, 42]. En 2004, Karikó et Weissman ont rapporté que l’ARNm transcrit in vitro contenait des contaminants d’ARNdb capables d’activer TLR3, induisant une réponse cytokinique [43]. Un autre indice important a été obtenu lorsque Koski, Karikó et Weissman, en collaboration avec Brian Czerniecki et ses collègues, ont démontré que la transfection de cellules dendritiques avec de l’ARNm transcrit in vitro stimulait une réponse cytokinique similaire à celle observée avec l’ARN procaryote. Des manipulations expérimentales visant à augmenter la longueur de la queue poly(A) de l’ARNm transcrit in vitro ont entraîné une réduction significative de la production d’IL-12. Cependant, cela n’expliquait pas entièrement les effets observés. Lorsque quatre homopolynucléotides, l’acide polyuridylique (pU), l’acide polyguanylique (pG), l’acide polycytidylique (pC) et l’acide polyadénylique (pA), ont été testés en utilisant l’IL-12 comme indicateur d’activation des cellules dendritiques, seul le pU a induit une réponse, suggérant que la composition nucléotidique jouait également un rôle [44]. Une découverte similaire, utilisant l’interféron alpha comme lecture, a été rapportée la même année par le groupe de Reis e Sousa dans leurs études sur la reconnaissance de l’ARN par TLR7 [41].
La percée de Kariko et Weissman
Karikó et Weissman ont poursuivi leurs études approfondies sur différents types d’ARN, travaux qui ont abouti à une publication majeure en 2005. Cette étude décrivait l’influence des modifications de bases de l’ARNm sur la réponse cytokinique des cellules dendritiques [45]. Ils ont montré que l’ARNm et l’ARNt eucaryotes, riches en modifications de bases, ne stimulaient pas de réponse cytokinique, contrairement à l’ARNm procaryote et à l’ARNm transcrit in vitro. Ils ont également montré que l’incorporation de pseudouridine (ψ), de 5-méthylcytidine (m5C), de N6-méthyladénosine (m6A), de 5-méthyluridine (m5U) ou de 2-thiouridine (s2U) dans l’ARNm transcrit in vitro abolissait l’activation des réponses inflammatoires lorsque ces ARNm étaient ajoutés aux cellules dendritiques [45]. L’incorporation de m6A et de s2U a quasiment aboli la reconnaissance par TLR3, tandis que l’activation de TLR7 et TLR8 a pu être évitée grâce à m6A, s2U, m5C, m5U et ψ. Il est important de noter que seules les modifications des uridines (m5U, s2U et ψ) ont aboli l’activation des cellules dendritiques (Figure 2).
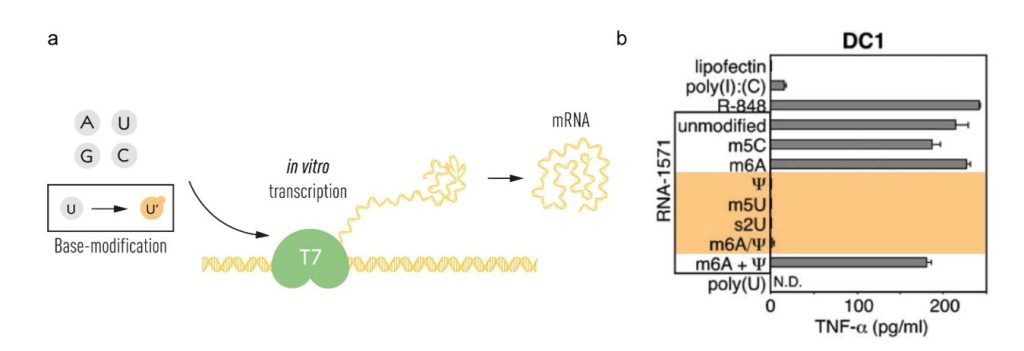
(a) Le système de transcription in vitro T7 a été utilisé pour produire de l’ARNm avec des bases canoniques (A, U, G et C) ou des bases modifiées. (b) Les bases utilisées pour la transcription in vitro de l’ARN-1571 sont indiquées, celles n’ayant pas induit la sécrétion de TNF-α étant représentées en orange (modifié d’après Karikó et al., Immunity 2005). © Comité Nobel de physiologie ou médecine. Ill. Mattias Karlén
À ce jour, les chercheurs ont identifié plus d’une centaine de modifications post-transcriptionnelles différentes de l’ARN et ont démontré que ces modifications sont plus fréquentes chez les eucaryotes que chez les procaryotes [46, 47]. La pseudouridine (Ψ) a été découverte dès 1951 [48] et figure parmi les modifications d’ARN les plus abondantes. Initialement présente dans les ARNt et les petits ARN nucléaires (snARN), elle a été plus récemment identifiée dans d’autres types d’ARN. Les cellules modifient l’ARN par des réactions enzymatiques ; par exemple, la synthèse de la pseudouridine est catalysée par les pseudouridine synthases, ou par des complexes de petites ribonucléoprotéines (snoRNP). Les modifications de l’ARN contribuent à sa stabilité, à la spécificité d’appariement des bases, à son repliement et à d’autres propriétés fonctionnelles. Parmi la centaine de modifications d’ARN connues [49], les données fonctionnelles restent limitées pour la plupart d’entre elles. Comprendre les implications physiologiques de ces modifications demeure donc un domaine de recherche actif.
La découverte de Karikó et Weissman a permis d’expliquer une observation faite plus de 40 ans auparavant par Isaacs et ses collègues, démontrant que l’introduction d’ARN désaminé dans les cellules induisait une réponse interféron de type I plus forte que l’ARN témoin [50]. La désamination augmente la proportion d’uridines dans l’ARN, dont Karikó et Weissman avaient démontré le rôle crucial dans l’activation des cellules dendritiques. Des travaux ultérieurs ont montré que l’utilisation de la N1-méthylpseudo-uridine (m1ψ), seule ou en combinaison avec la m5C, améliorait encore la plateforme d’ARNm, à la fois en réduisant la reconnaissance par les récepteurs de l’immunité innée et en augmentant l’expression protéique [51]. Cette dernière amélioration s’explique en partie par une occupation ribosomique accrue sur l’ARNm contenant la m1ψ [52]. Aujourd’hui, la m1ψ est la base modifiée la plus couramment utilisée dans la production de vaccins à ARNm, notamment dans les deux vaccins contre la COVID-19 approuvés fin 2020, comme nous le verrons plus loin.
Suite à leur découverte majeure selon laquelle l’incorporation de bases modifiées permet d’éviter l’activation immunitaire indésirable induite par l’ARNm transcrit in vitro, Karikó et Weissman ont démontré que l’ARNm contenant de la pseudouridine était également traduit plus efficacement, entraînant une production protéique accrue dans les cellules ayant internalisé cet ARNm [53] (Figure 3). Dans la même étude, ils ont montré que l’administration d’ARNm modifié dans la rate de souris induisait une augmentation de la production protéique et une diminution de l’activation immunitaire, une démonstration importante pour de futures applications thérapeutiques. Karikó, Weissman et leurs collègues ont par ailleurs démontré que l’ARNm transcrit in vitro active la protéine kinase R (PKR), une protéine antivirale qui protège les cellules des agents pathogènes en reconnaissant l’ARNdb par phosphorylation du facteur d’initiation de la traduction eucaryote 2 alpha (eIF2α), bloquant ainsi la traduction protéique. L’équipe a montré que l’utilisation de bases modifiées réduisait l’activation de la PKR et améliorait la production protéique [54]. La reconnaissance de l’ARNm transcrit in vitro par l’oligoadénylate synthétase 2’5’ (OAS) et la dégradation par l’enzyme Rnase L induite par l’OAS ont également diminué avec l’ARN contenant des bases modifiées [55].
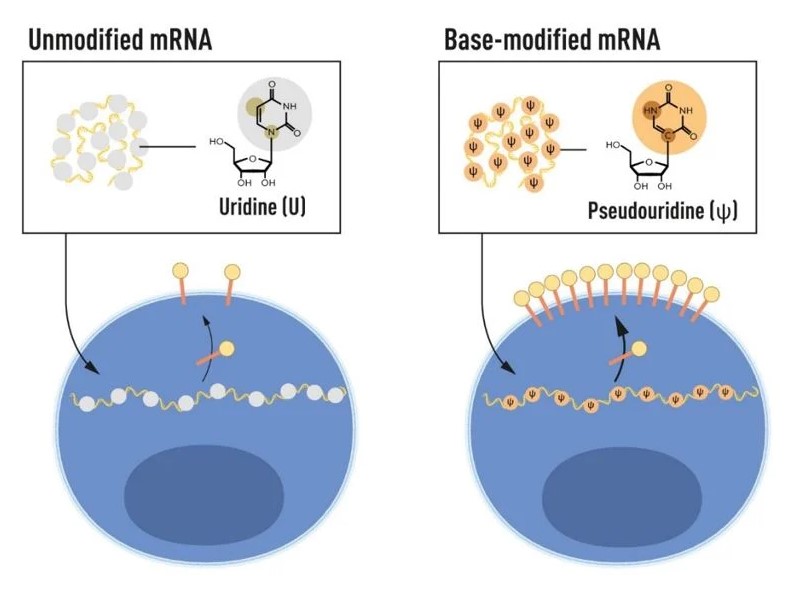
Cet ARNm modifié a été produit en substituant les uridines (U) par de la pseudouridine (Ψ). L’introduction de cet ARNm modifié dans les cellules a induit une production protéique supérieure à celle obtenue avec l’ARNm non modifié. © Comité Nobel de physiologie ou médecine. Ill. Mattias Karlén
De plus, Karikó et ses collègues ont montré que les contaminants dsRNA produits lors de la transcription in vitro pouvaient être éliminés par une étape de purification HPLC [56], ou comme rapporté plus tard avec Uğur Şahin et ses collègues chez BioNTech, en utilisant une étape de purification à base de cellulose [57], améliorant ainsi l’expression des protéines à partir de l’ARNm transcrit in vitro.
Recherches préliminaires aux vaccins à ARN messager contre la COVID-19
En 2010, trois grandes entreprises, dont les programmes étaient axés sur la technologie émergente de l’ARNm, avaient été créées : CureVac, fondée en 2000, visait à développer des vaccins contre les infections et le cancer ; BioNTech, fondée en 2008, avait pour objectif de développer des vaccins anticancéreux personnalisés ; et Moderna, fondée en 2010, prévoyait d’utiliser la plateforme ARNm pour reprogrammer des cellules somatiques en cellules pluripotentes et administrer des protéines thérapeutiques, par exemple pour réparer les tissus endommagés. Ces trois entreprises ont collaboré étroitement avec des chercheurs universitaires afin d’améliorer la technologie et d’évaluer leurs plateformes respectives dans des domaines pathologiques d’intérêt.
L’équipe de CureVac, composée notamment d’Ingmar Hoerr, Günter Jung, Steve Pascolo et Hans-Georg Rammensee, avait très tôt perçu le potentiel de la technologie ARNm. Elle a développé des approches pour améliorer l’efficacité de la production de protéines grâce à l’optimisation des régions non traduites 5′ et 3′ de l’ARNm et à l’optimisation des codons, sans utiliser de bases modifiées. En 2000, ils ont rapporté que l’administration d’ARN, nu ou complexé à des liposomes, induisait des réponses immunitaires adaptatives spécifiques à l’antigène chez la souris (réponses anticorps et lymphocytes T CD8+), l’ARN encapsulé dans des liposomes induisant des réponses plus importantes [58]. Environ huit ans plus tard, ils ont évalué leur premier vaccin à ARNm chez l’humain, en extrayant du matériel génétique de tumeurs de patients atteints de mélanome. Ce matériel a servi à générer un ARNm administré comme vaccin autologue, avec le facteur de croissance des granulocytes-macrophages (GM-CSF) comme adjuvant. Cette approche s’est avérée sûre et a permis d’accroître les réponses immunitaires antitumorales chez certains patients [59]. En 2012, l’équipe de CureVac a rapporté l’induction de réponses immunitaires protectrices contre l’infection par le virus de la grippe dans plusieurs modèles animaux [60]. En 2017, le premier vaccin à ARNm contre une maladie infectieuse, la rage, a été testé lors d’essais cliniques.
Les activités dans le domaine des vaccins à ARNm connaissent depuis un essor rapide. En 2017, Norbert Pardi et Weissman [61] ainsi que Michael Diamond et ses collègues de la faculté de médecine de l’université Washington [62] ont rapporté des résultats précliniques prometteurs concernant des vaccins contre le virus Zika à ARNm utilisant des bases modifiées. Cette dernière étude, portant sur la vaccination de femmes enceintes, a démontré une protection contre la transmission du virus au fœtus, une préoccupation majeure liée aux infections par le virus Zika. Toujours en 2017, Moderna a annoncé le lancement d’un essai clinique avec un vaccin à ARNm contre le virus Zika. Moderna a également initié deux essais cliniques de phase I afin d’évaluer l’innocuité et l’immunogénicité de ses candidats vaccins à ARNm contre les virus de la grippe H10N8 et H7N9, deux souches de grippe aviaire à potentiel pandémique [63, 64].
Au moment même où se déroulaient les essais cliniques du vaccin contre le Zika, Moderna a également entamé une collaboration avec Barney Graham et son équipe du Centre de recherche sur les vaccins des NIH afin de développer un vaccin à ARNm contre le coronavirus du syndrome respiratoire du Moyen-Orient (MERS-CoV). Ce vaccin codait une forme stabilisée en phase de préfusion de la protéine Spike du MERS, où, entre autres modifications, des prolines ont été introduites dans le domaine S2 pour empêcher la transition de la forme métastable de préfusion vers la forme de post-fusion [65]. Des travaux préliminaires de Qiao et al. avaient montré que l’introduction de prolines dans le domaine HA2 de l’hémagglutinine du virus de la grippe, qui subit une transition boucle-hélice à pH faible, perturbe la capacité du virus de la grippe à fusionner avec les membranes de l’hôte [66]. Partant de ce constat, et sachant que des virus de différentes familles ont développé des mécanismes similaires de fusion avec les cellules cibles, des prolines positionnées de manière appropriée ont été insérées dans les glycoprotéines de spicule de plusieurs virus afin de les stabiliser dans leur conformation de préfusion respective, notamment pour le VIH-1 [67], le virus respiratoire syncytial [68] et le SARS-CoV-2 [69]. La structure à haute résolution de la protéine de spicule du SARS-CoV-2, publiée en un temps record par l’équipe de Jason McLellan début 2020, s’est révélée inestimable pour plusieurs vaccins efficaces contre la COVID-19, ainsi que pour la définition des épitopes d’anticorps neutralisants et des mutations d’échappement immunitaire chez les variants du SARS-CoV-2 apparus ultérieurement. Ces informations sont cruciales pour notre compréhension de la protection immunitaire induite par la vaccination. La forme stabilisée en préfusion de la protéine Spike du SARS-CoV-2 a été utilisée dans les vaccins à ARNm développés par Pfizer/BioNTech et Moderna (figure 4), ainsi que dans le vaccin à vecteur de Janssen et le vaccin à base de protéines développé par Novavax.
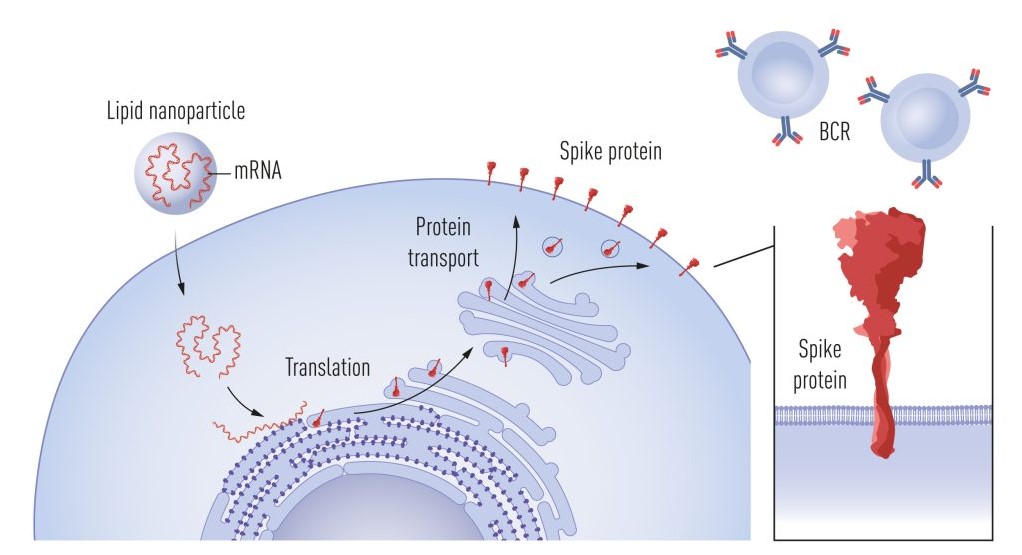
Après l’internalisation de l’ARNm dans les cellules, facilitée par des nanoparticules lipidiques, l’ARNm sert de matrice pour la production de la protéine Spike. La protéine Spike est alors exprimée transitoirement à la surface cellulaire, où elle est reconnue par les lymphocytes B via leurs récepteurs (BCR), stimulant ainsi la sécrétion d’anticorps spécifiques de la protéine Spike. © Comité Nobel de physiologie ou médecine. Ill. Mattias Karlén
Le moment était propice aux vaccins à ARN messager.
Lorsque la pandémie a éclaté début 2020, les entreprises spécialisées dans l’ARNm ont réagi rapidement pour développer des vaccins contre la COVID-19. BioNTech et Moderna ont opté pour l’ARNm à bases modifiées, s’appuyant sur les découvertes de Karikó et Weissmann. BioNTech, sous la direction d’Uğur Şahin et d’Özlem Türeci, a travaillé en partenariat avec Pfizer [70, 71], tandis que Moderna a collaboré étroitement avec le VRC/NIH, où Barney Graham et son équipe ont réalisé l’évaluation du vaccin [72-74]. Sans aucun doute, la pandémie de SARS-CoV-2 a été un événement décisif qui a entraîné des investissements massifs dans la technologie des vaccins à ARNm, notamment la conception d’essais cliniques menés en parallèle plutôt que séquentiellement, réduisant considérablement la durée des essais tout en permettant de mener à bien toutes les étapes nécessaires [75]. Le financement et le soutien collectifs des gouvernements, des organisations internationales et de l’industrie ont permis de mener à bien les essais d’innocuité et d’efficacité des vaccins en un temps record. Les vaccins à ARNm de Pfizer/BioNTech et de Moderna ont tous deux obtenu l’autorisation de mise sur le marché moins d’un an après l’apparition du SARS-CoV-2. Ce développement a été rendu possible grâce à des décennies de recherche fondamentale et d’optimisation des plateformes d’ARNm, comme décrit dans [76]. Les vaccins à ARNm de Pfizer/BioNTech et de Moderna présentaient tous deux une substitution complète de l’uridine par la N1-méthylpseudouridine (m1ψ) afin d’éviter les réactions inflammatoires indésirables, d’accélérer la traduction des protéines et de permettre l’utilisation de quantités plus importantes d’ARNm dans chaque dose vaccinale.
Les essais de phase 3, basés sur les résultats obtenus après deux vaccinations à ARNm, ont démontré un niveau de protection très élevé contre la COVID-19 symptomatique : 95 % d’efficacité pour le vaccin de Pfizer/BioNTech (Polack 2020) et 94 % pour celui de Moderna (Baden 2021). Les deux vaccins ont induit de fortes réponses anticorps, ainsi que des réponses des lymphocytes B et T mémoire, offrant une protection contre les formes graves de la maladie et le décès. Des études de suivi ont montré que les réponses sérologiques étaient relativement transitoires, et la communauté scientifique a rapidement démontré que des injections de rappel supplémentaires amélioraient considérablement la protection, notamment contre le variant Omicron, plus infectieux [77]. La propagation de nouveaux variants du SARS-CoV-2 est bien documentée et plusieurs sous-variants Omicron circulent actuellement. La communauté scientifique internationale continue de surveiller l’évolution du virus afin de suivre l’émergence de nouveaux variants et d’orienter la conception de vaccins mis à jour. L’année écoulée a démontré que la plateforme ARNm se prête à la production de vaccins mis à jour à une vitesse actuellement inégalée par les autres plateformes vaccinales.
Plusieurs vaccins contre la COVID-19 ont contribué à sauver des vies et à réduire la pression insoutenable sur les systèmes de santé depuis 2021. La technologie ARNm représente un atout essentiel parmi les plateformes utilisables pour la production de vaccins, notamment en cas de pandémie, lorsque l’extensibilité et la flexibilité sont primordiales. L’utilisation généralisée des deux vaccins à ARNm contre la COVID-19 au cours des dernières années démontre le potentiel important de la technologie et montre que les effets indésirables graves des deux vaccins à ARNm homologués étaient exceptionnellement rares [78], fournissant une base solide pour les applications futures.
Les bases modifiées sont-elles nécessaires pour toutes les applications cliniques de l’ARN messager ?
Plusieurs applications de la plateforme ARNm sont actuellement en développement, notamment pour les vaccins contre les infections et le cancer, ainsi que pour l’administration de protéines thérapeutiques ou immunomodulatrices. Les exigences relatives aux bases modifiées peuvent varier selon les applications de l’ARNm. Pour les vaccins prophylactiques administrés à un grand nombre de personnes en bonne santé, la réactogénicité à l’injection est un facteur important. Une réaction transitoire légère peut être acceptable si elle est limitée au site d’injection, tandis que des symptômes inflammatoires systémiques tels que fièvre, myalgies et céphalées sont indésirables, voire inacceptables selon leur gravité. Le niveau de réactogénicité acceptable doit être déterminé pour chaque vaccin, en fonction de l’importance du bénéfice lié à l’induction d’une réponse protectrice. Trouver le juste équilibre entre réactogénicité et efficacité pour un vaccin donné peut donc s’avérer complexe [79].
Khoury et al. ont rapporté que les effets protecteurs de tous les vaccins contre la COVID-19 pour lesquels des résultats étaient disponibles à la mi-2021 étaient corrélés aux titres moyens d’anticorps neutralisants contre le virus fondateur, obtenus dans chacun des essais [80]. Ces résultats concordent avec les données d’autres vaccins antiviraux homologués, pour lesquels la protection contre la maladie est corrélée à la présence d’anticorps neutralisants [81]. Une fois disponibles les résultats de l’essai clinique de CureVac [82], ils ont été comparés à ceux d’autres essais [83]. Cette analyse a montré que les titres d’anticorps neutralisants induits par le vaccin CureVac étaient inférieurs à ceux induits par les vaccins à ARNm de Pfizer/BioNTech et de Moderna, suggérant que la dose d’ARNm plus faible utilisée dans l’essai de CureVac a compromis l’effet protecteur de ce vaccin. Une comparaison définitive des différents vaccins à ARNm est complexifiée par la circulation de variants plus résistants à la neutralisation au moment où CureVac a mené son essai de phase 3. Cependant, les résultats confirment que l’utilisation de modifications de bases dans l’ARNm transcrit in vitro codant pour la protéine Spike du SARS-CoV-2 était essentielle au développement de vaccins à ARNm pouvant être administrés à des doses suffisamment élevées pour protéger contre la COVID-19.
À l’avenir, d’autres approches visant à optimiser l’ARNm pour le développement de produits cliniquement utiles, comme l’ARN circulaire [84], les réplicons [85] et d’autres types d’ARN sans modifications de bases, seront probablement mises au point. Des études cliniques récentes, associant une vaccination thérapeutique par ARNm à l’inhibition des points de contrôle immunitaire à l’aide d’antigènes tumoraux mutés ou non mutés, ont démontré l’induction réussie de réponses des lymphocytes T spécifiques de la tumeur chez des patients atteints de mélanome et d’adénocarcinome canalaire pancréatique [86-88]. L’ARNm utilisé dans ces essais comportait des bases non modifiées, mais comportait des modifications de la queue poly(A) décrites pour accroître la stabilité et l’efficacité de la traduction de l’ARNm [89]. Ainsi, des approches alternatives pour générer des vaccins et des thérapies efficaces à base d’ARNm sont en cours de développement. De nombreux essais cliniques utilisent actuellement différentes formes d’ARNm pour induire des réponses prophylactiques ou thérapeutiques dans les domaines des infections [90] et du cancer [91, 92], et leur nombre devrait augmenter dans les années à venir.
Résumé
L’approbation, fin 2020, de deux vaccins à ARNm efficaces et sûrs contre la COVID-19 a propulsé le domaine des vaccins à ARNm dans une nouvelle ère. La découverte que l’utilisation de bases modifiées dans l’ARNm transcrit in vitro permet de contourner les réponses inflammatoires indésirables et d’accroître la production de protéines après son administration aux cellules démontre l’importance de la recherche fondamentale. Les résultats publiés par Karikó et Weissman dans leur article fondateur de 2005, bien que peu remarqués à l’époque, ont jeté les bases d’avancées cruciales qui ont été d’un grand secours pour l’humanité durant la pandémie de COVID-19.
Gunilla Karlsson Hedestam, PhD, Professeure à l’Institut Karolinska (Gunilla.Karlsson.Hedestam@ki.se), Membre du Comité Nobel
Rickard Sandberg, PhD, Professeur à l’Institut Karolinska (Rickard.Sandberg@ki.se), Membre du Comité Nobel
Illustrations : Mattias Karlén